GBIF: cartografía de la distribución de especies ¶
La Infraestructura Mundial de Información en Biodiversidad GBIF integra información cartográfica con datos de biodiversidad a nivel mundial. Actualmente están integrados 53 países en todo el mundo así como 43 organizaciones internacionales bajo el objetivo de proporcionar un acceso libre a la información vinculada con la biodiversidad.(1)
Es decir que nuestros amigos de GBIF, nos permiten consultar registros de observación de Especies disponibles en las principales colecciones naturales de los paises bajo un esquema (2) que bien nos señala @daiesco
The schema in GBIF Colombia 🇨🇴 (pic.twitter.com/xTGRxbqjRb
— Dairo Escobar (@daiesco) March 5, 2020
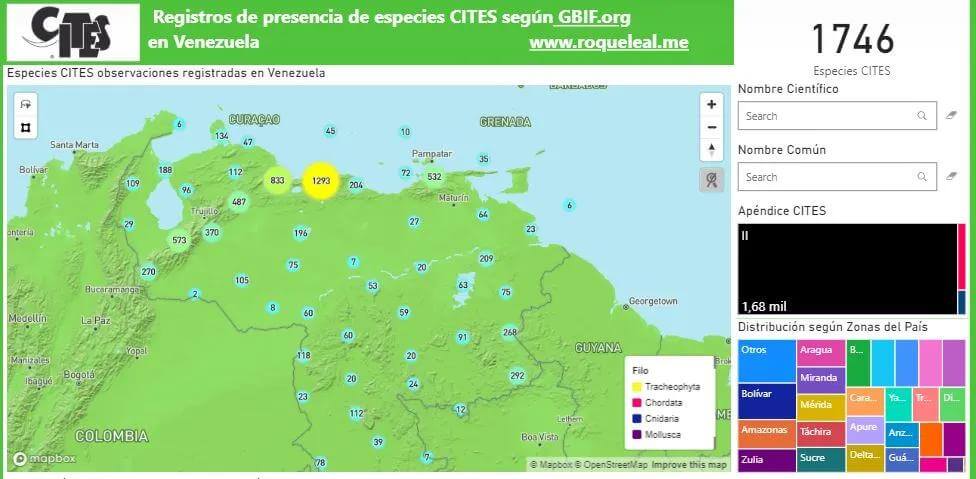
Latinoamérica dispone de un gran riqueza en biodiversidad, razón por la cual es oportuna la ocasión de realizar un ejercicio práctico para contar una historia; en este caso de las Especies protegidas bajo la Convención sobre el Comercio Internacional de Especies Amenazadas de Fauna y Flora Silvestres CITES (3) utilizando como referencia el listado de Especies asociado a Venezuela, el cual puede consultaremos usando el API de SPECIES+/CITES CHECKLIST API (4)

Venezuela además del petróleo, ocupa el séptimo lugar dentro de los países con mayor biodiversidad del mundo (5) por lo que analizar la distribución de sus Especies protegidas nos puede dar un vistazo más definido sobre la magnitud de la biodiversidad venezolana además de su distribución espacial utilizando estas dos valiosas fuentes de datos, basado en el script de consulta Python desarrollado por Marie Grosjean (6)
Fuente de los Datos¶
Se necesitarán las siguientes fuentes de datos para extraer y generar la información requerida:
1.- Consultar el GBIF API utilizando nuestras credenciales. (1)
2.- Lista de Especies protegidas bajo el Convenio CITES para Venezuela. (4)
3.- Script de Funciones para la consulta del GBIF API de consulta Python. (6)
Los datos se utilizarán en los siguientes procesos:
1- Para descubrir la distribución espacial y datos de observación de las especies.
2- Para identificar las Especies CITES en Venezuela.
3- Para crear los query's de consultas.
Librerias¶
#Libreria de pandas
import pandas as pd
#Libreria de kepler-gl
from keplergl import KeplerGl
# Scripts de funciones https://github.com/ManonGros/Small-scripts-using-GBIF-API/blob/master/query_species_list/functions_query_from_species_list.py
from functions_query_from_species_list import *
Constantes¶
# Credenciales para el API de GBIF
GBIF_USER_NAME = "xxx"
GBIF_PASSWORD = "xxx"
GBIF_NOTIFICATION_ADDRESSES = "xxx"
GBIF_DOWNLOAD_FORMAT = "SIMPLE_CSV"
# Directorio de entrada
INPUT_DIR = "C:/Users/xxx/"
# Archivo CSV de entrada con lista de especies a procesar tomado de la Lista de Especies protegidas bajo el Convenio CITES para Venezuela. (4)
INPUT_CHECKLIST = INPUT_DIR + "cites_vzla.csv"
# Columna con el nombre científico de la especie
INPUT_SCINAME_COL = "full_name"
# Directorio de salida
OUTPUT_DIR = "C:/Users/xxx/"
# Archivo CSV con especies del archivo de entrada que se procesan
INPUT_CHECKLIST_PROCESSED_SPECIES = OUTPUT_DIR + "especies-procesadas.csv"
# Archivo CSV con especies del archivo de entrada que no se procesan
INPUT_CHECKLIST_NON_PROCESSED_SPECIES = OUTPUT_DIR + "especies-no-procesadas.csv"
# Archivo CSV de salida con los registros de presencia de todas las especies
OCCURRENCES_CSV = INPUT_DIR + "especies-procesadas.csv"
# Número máximo de registros a desplegar en los dataframes de Pandas
pd.options.display.max_rows = 5
Carga de datos¶
# Carga del archivo CSV de entrada (Lista de Especies CITES) en un dataframe de Pandas
input_species_df = pd.read_csv(INPUT_CHECKLIST, encoding='utf_8')
input_species_df
| id | kingdom_name | phylum_name | class_name | order_name | family_name | genus_name | species_name | subspecies_name | full_name | ... | synonyms_with_authors | english_names | spanish_names | french_names | countries_iso_codes | countries_full_names | current_listing | current_parties_iso_codes | current_parties_full_names | current_listing_changes | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 82867 | Plantae | NaN | NaN | Orchidales | Orchidaceae | Epidendrum | agathosmicum | NaN | Epidendrum agathosmicum | ... | [List] | [List] | [List] | [List] | [List] | [List] | II | [List] | [List] | [List] |
| 1 | 81567 | Plantae | NaN | NaN | Orchidales | Orchidaceae | Epidendrum | alpicola | NaN | Epidendrum alpicola | ... | [List] | [List] | [List] | [List] | [List] | [List] | II | [List] | [List] | [List] |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 1744 | 4281 | Animalia | Chordata | Aves | Apodiformes | Trochilidae | Topaza | pella | NaN | Topaza pella | ... | [List] | [List] | [List] | [List] | [List] | [List] | II | [List] | [List] | [List] |
| 1745 | 5338 | Animalia | Chordata | Aves | Apodiformes | Trochilidae | Topaza | pyra | NaN | Topaza pyra | ... | [List] | [List] | [List] | [List] | [List] | [List] | II | [List] | [List] | [List] |
1746 rows × 23 columns
Tenemos para Venezuela 1746 Especies protegidas por el CITES
# Se obtienen las llaves de las especies a través del API de GBIF
gbif_species_df = match_species(input_species_df, INPUT_SCINAME_COL)
gbif_species_df
# Se construye una lista de nombres que van a procesarse
gbif_species_processed_df = gbif_species_df.loc[(gbif_species_df["matchType"]=="EXACT")]
gbif_species_processed_df.to_csv(INPUT_CHECKLIST_PROCESSED_SPECIES)
gbif_species_processed_df
# Se construye una lista de nombres que no van a procesarse
gbif_species_non_processed_df = gbif_species_df.loc[~((gbif_species_df["matchType"]=="EXACT"))]
gbif_species_non_processed_df.to_csv(INPUT_CHECKLIST_NON_PROCESSED_SPECIES)
gbif_species_non_processed_df
| canonicalName | class | classKey | confidence | family | familyKey | genus | genusKey | inputName | kingdom | ... | orderKey | phylum | phylumKey | rank | scientificName | species | speciesKey | status | synonym | usageKey |
|---|
0 rows × 24 columns
# Se separa la lista de llaves
key_list = gbif_species_processed_df.usageKey.tolist()
key_list
[5959225]
Aqui construiremos la consula que nos permitirá descargar el listado de las Especies y sus datos comprimidos en un ZIP y que podremos descargar desde el modulo de descargas del portal de GBIF dentro de la cuenta del usuario
# Se construye una consulta para descarga en el portal de GBIF
download_query = {}
download_query["creator"] = GBIF_USER_NAME
download_query["notificationAddresses"] = [GBIF_NOTIFICATION_ADDRESSES]
download_query["sendNotification"] = True
download_query["format"] = GBIF_DOWNLOAD_FORMAT
download_query["predicate"] = {"type":"and", "predicates":
[
{"type":"equals", "key":"HAS_COORDINATE", "value":"true"},
{"type":"equals", "key":"HAS_GEOSPATIAL_ISSUE", "value":"false"},
{"type":"in", "key": "TAXON_KEY", "values":key_list},
{
"type": "or",
"predicates": [
{
"type": "equals",
"key": "COUNTRY",
"value": "VE"
}
]
}
]
}
download_query
{'creator': 'roqueleal',
'notificationAddresses': ['roqueleal@gmail.com'],
'sendNotification': True,
'format': 'SIMPLE_CSV',
'predicate': {'type': 'and',
'predicates': [{'type': 'equals', 'key': 'HAS_COORDINATE', 'value': 'true'},
{'type': 'equals', 'key': 'HAS_GEOSPATIAL_ISSUE', 'value': 'false'},
{'type': 'in', 'key': 'TAXON_KEY', 'values': [5959225]},
{'type': 'or',
'predicates': [{'type': 'equals', 'key': 'COUNTRY', 'value': 'VE'}]}]}}
# Consulta para el API de GBIF
create_download_given_query(GBIF_USER_NAME, GBIF_PASSWORD, download_query)
# Respuesta esperada:
# ok
# <Response [201]>
ok
<Response [201]>
La consulta al API genera un archivo ZIP para descarga en https://www.gbif.org/user/download
Este dataset son los resulados de las consultas del API de GBIF
occurrences_df = pd.read_csv(OCCURRENCES_CSV, sep='\t')
occurrences_df
C:\Program Files (x86)\Microsoft Visual Studio\Shared\Anaconda3_64\lib\site-packages\IPython\core\interactiveshell.py:2785: DtypeWarning: Columns (10,14,18,39,40,41,43,45,46,48) have mixed types. Specify dtype option on import or set low_memory=False. interactivity=interactivity, compiler=compiler, result=result)
| gbifID | datasetKey | occurrenceID | kingdom | phylum | class | order | family | genus | species | ... | identifiedBy | dateIdentified | license | rightsHolder | recordedBy | typeStatus | establishmentMeans | lastInterpreted | mediaType | issue | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1211815285 | c5c4a23e-2035-4416-ab64-032d6df52ddb | URI:catalog:ROM:Mammals:107836 | Animalia | Chordata | Mammalia | Carnivora | Canidae | Cerdocyon | Cerdocyon thous | ... | NaN | NaN | CC_BY_NC_4_0 | Royal Ontario Museum; ROM | Lim, BK; Lee, Te, Jr; Hanson, Jd | NaN | NaN | 2020-02-10T20:50:01.515Z | NaN | COORDINATE_ROUNDED;GEODETIC_DATUM_ASSUMED_WGS84 |
| 1 | 1453241628 | 50c9509d-22c7-4a22-a47d-8c48425ef4a7 | http://www.inaturalist.org/observations/4634444 | Animalia | Chordata | Reptilia | Squamata | Boidae | Eunectes | Eunectes murinus | ... | NaN | 2016-11-25T13:09:39Z | CC_BY_NC_4_0 | Brad Walker | Brad Walker | NaN | NaN | 2020-02-19T16:22:29.228Z | StillImage;StillImage;StillImage;StillImage | GEODETIC_DATUM_ASSUMED_WGS84 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 139875 | 1317757887 | 821cc27a-e3bb-4bc5-ac34-89ada245069d | http://n2t.net/ark:/65665/3188edd67-eb6a-4a72-... | Animalia | Chordata | Mammalia | Rodentia | Cuniculidae | Cuniculus | Cuniculus paca | ... | NaN | NaN | CC0_1_0 | NaN | Smithsonian Venezuelan Project | NaN | NaN | 2020-02-13T22:08:24.058Z | NaN | GEODETIC_DATUM_ASSUMED_WGS84 |
| 139876 | 1998558297 | 861e6afe-f762-11e1-a439-00145eb45e9a | 39f2b678-cec7-4e16-b3e6-ba9a8c310879 | Plantae | Tracheophyta | Liliopsida | Asparagales | Orchidaceae | Epidendrum | Epidendrum alsum | ... | Dunsterville | NaN | CC_BY_4_0 | President and Fellows of Harvard College | J. A. Steyermark & C. Brewer-Carias | NaN | NaN | 2020-02-11T00:11:35.141Z | NaN | GEODETIC_DATUM_ASSUMED_WGS84;GEODETIC_DATUM_IN... |
139877 rows × 50 columns
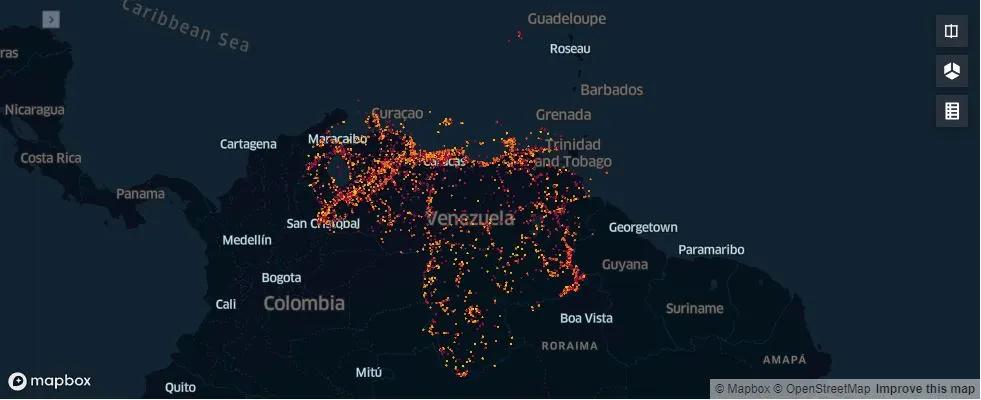
Tenemos un Total de 139.877 registros de observaciones de Especies protegidas por CITES para Venezuela
Visualización¶
Vamos a utilizar kepler.gl para visualizar estos datos, igualmente una visualización más completa esta disponible en mi web www.roqueleal.me
vzla_cites = KeplerGl()
vzla_cites.add_data(data=occurrences_df, name='data_1')
vzla_cites
User Guide: https://github.com/keplergl/kepler.gl/blob/master/docs/keplergl-jupyter/user-guide.md
KeplerGl(data={'data_1': {'index': [0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, …